Basic Info of miRNA
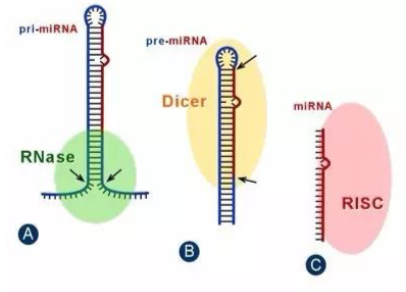
pri-miRNA:长度从几百到几千个碱基不等,带有 5‘帽子和 3’polyA 尾巴,以及 1 到数个发 夹径环结构。
pre-miRNA:pri-miRNA 在核酸酶 Drosha 和其辅助因子 Pasha 的作用下被处理成 70 个核 苷酸组成的前体。
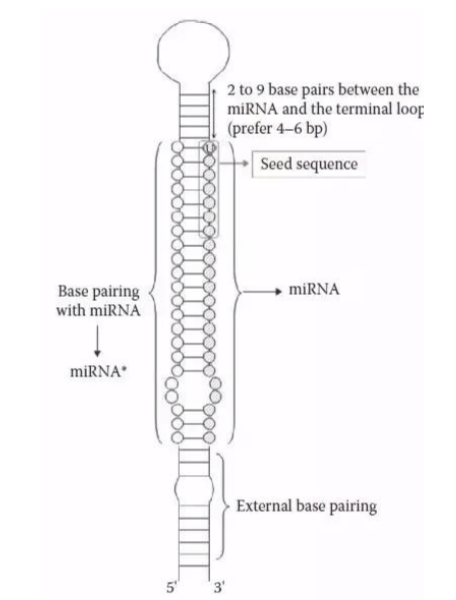
mature miRNA:pre-miRNA 经进一步经过 DICER 酶剪切,形成长度约为 22 个碱基的单 链成熟 miRNA。
miRNA 种子区(“seed”region)
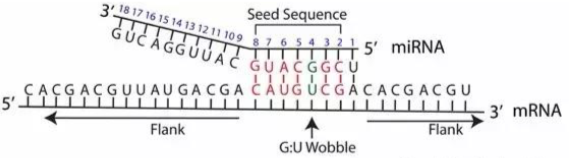
5’端 2-8 个碱基被称为种子序列, 是miRNA的保守区域, 跟mRNA 3’UTR 结合行使调控功能。
miRNA命名规则
1 确定命名规则之前发现的 miRNA(microRNA),则保留原来名字,如 hsa-let-7.
2 miRNA(microRNA)成熟体简写成 miR,再根据其物种名称,及被发现的先后顺序加上阿拉伯数字,如 hsa-miR-122;
3 高度同源的 miRNA(microRNA)在数字后机上英文小写字母(a,b,c,…),如 hsa-miR-34a,hsa-miR-34b,hsa-miR-34c 等;
4 由不同染色体上的 DNA 序列转录加工而成的具有相同成熟体序列的 miRNA(microRNA),则在后面加上阿拉伯数字以示区分,如hsa-miR-199a-1 和 hsa-miR- 199a-2;
5 通常一个 miRNA(microRNA)前体长度大约为 70~80nt,很可能两个臂分别产生 miRNA(microRNA)。以前的做法是:表达水平较高的 miRNA(microRNA)后面不加任何符号,而表达水平较低的 miRNA(microRNA)后面加上号,如 rno-miR-9。有时带“*”的 miRNA(microRNA)就根本 不出现。而如果没有明显的表达差异,则以“-5p”和“-3p”分别命名。如 hsa-miR-26b-5p 和 hsa-miR- 26b-3p,分别表明从 hsa-mir-26b 前体的 5’端臂和 3’端臂加工而来的。
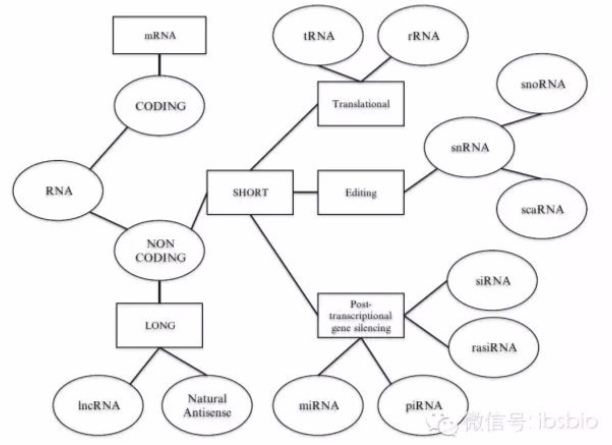
RNA分类树
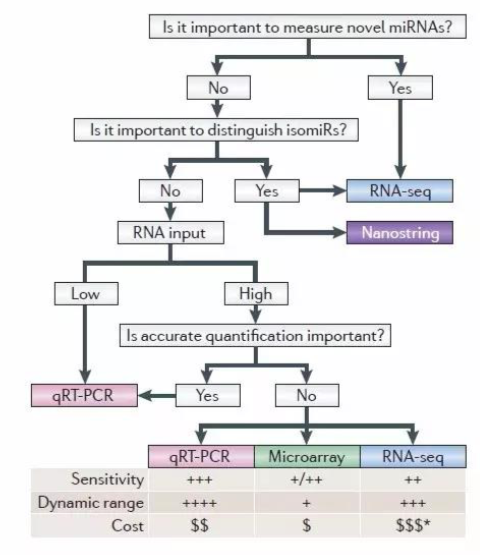
RNA筛选平台的选择
Databases related to miRNA
Here is a list of miRNA related Database, including ‘miRNA expression’, ‘Target Expression’, ‘Transcription Factor-miRNA Regulation’, ‘Experimental Target’, ‘Diseases/Pathways’, ‘miRNA-related SNP information’, ‘miRNA basic Information’, ‘miRNA-ncRNA Interaction’, ‘Literature Mining’ and ‘Predicted Target’.
Today, I will focus on miRNA basic Information.
miRBase / isomiR Bank / miRNA isomir database
miRBase (Release 22.1: October 2018)
Info: 38589 entries
miRBase 记录了 miRNA(microRNA)前体序列及 miRNA 成熟体序列:
miRNA(microRNA)前体
发夹状结构的 miRNA(microRNA)前体转录本以“mir”命名,其编号以“MI”编号,如人的 miRNA 122 的前体 ID 为hsa-mir-122,Accession 为 MI0000442。
miRNA(microRNA)成熟体
大约 20~23nt 的 miRNA(microRNA)成熟体以“miR”命名,其编号以“MIMAT”编号,如 人的 miR-122 有两个成熟体,其中之一 ID为 hsa-miR-122-5p ,Accession为 MIMAT0000421;另一个为 ID 为 hsa-miR-122-3p ,Accession 为 MIMAT000 4590。
Download:
1.mature.fa -> miR (from human)
- length range :16-35 base, but most of it center at( 17-26 ), which frequency is bigger than 100.
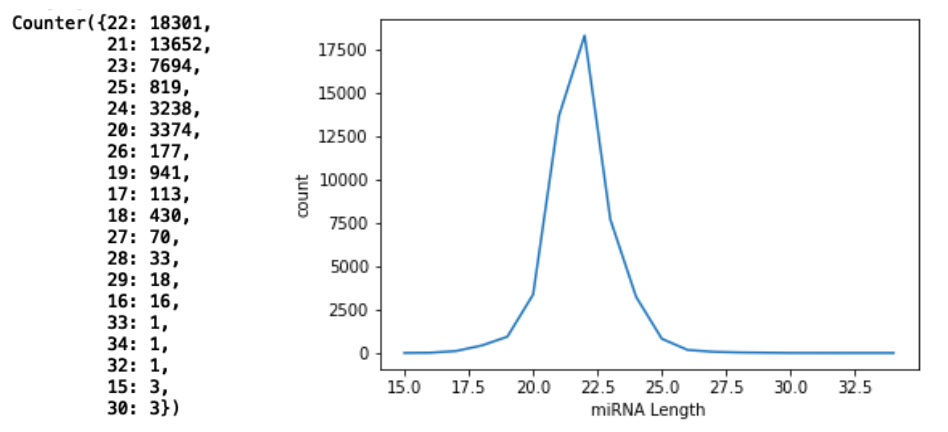
Script for statistics and plot
import numpy as np
import collections
import matplotlib.pyplot as plt
#统计数据种类
data = np.loadtxt('mature_len.txt')
uniq = np.unique(data)
#读入每条miRNA的长度
lst = []
with open('mature_len.txt','r') as f:
for line in f:
lst.append(int(line))
#统计频数
d = collections.Counter(lst)
#存入list,划线
length = []
freq = []
for i in uniq:
key = int(i)
length.append(int(i))
freq.append(d[key])
plt.plot(length,freq)
plt.xlabel('miRNA Length')
plt.ylabel('count')
plt.show()
2.hairpin.fa
IsomiR Bank
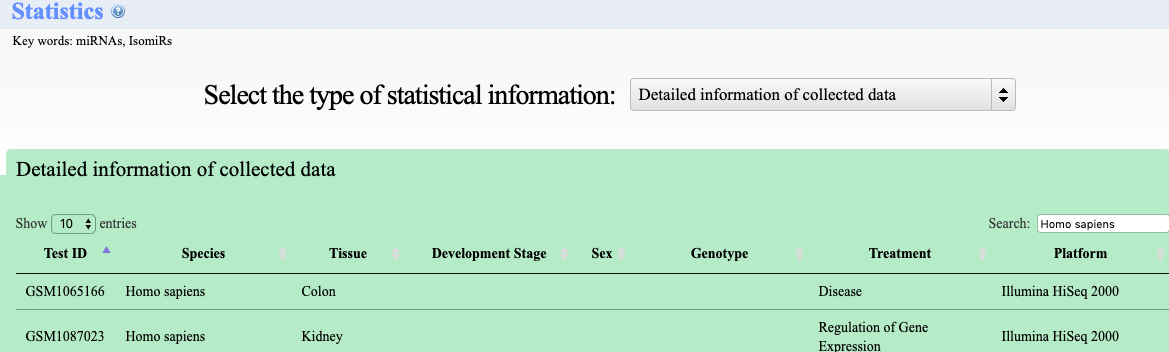
Info: 2,727 samples (Small RNA NGS data downloaded for ArrayExpress) from eight species (Arabidopsis thaliana, Drosophila melanogaster, Danio rerio, Homo sapiens, Mus musculus, Oryza sativa, Solanum lycopersicum and Zea mays) were analyzed by using CPSS. We have stored 308,919 isomiRs from 4,706 mature miRNAs in our IsomiR Bank database.
In this Database, you can find:
- related raw sequence dataset list in Statistics as belowed. eg, SRR764457
miRNA isomir database
Ref. 2010 - Complexity of the microRNA repertoire revealed by next-generation sequencing
Raw Data Preprocess Script
Seed Region of microRNA
种子区(seed region)指的是miRNA上进化最为保守的片段,从第2个到第8个核苷酸,通常与mRNA 3’-UTR上的靶位点完全互补。